Главная | О сайте | Задачи | Проекты | Результаты | Диверсификация | Новости | Вопросы | История | Информация | Ссылки
Космическая погода на текущий час
Войти / Регистрация
Секция Совета РАН по космосу
Наноструктуры из ДНК можно собирать по принципу конструктора «Лего»
Всего несколько дней назад на «Элементах» появилась новость Елены Наймарк о том, что с помощью методики ДНК-оригами удалось получить действующий ионный канал. Новость заканчивалась фразой о том, что наука в этой области движется бешеными темпами, и даже работы двух-трехлетней давности считаются здесь «прошлым». И вот, дорогие читатели, перед вами яркая иллюстрация этой фразы: в последнем номере Science предложена новая методика получения сложных трехмерных структур из ДНК. Сборка ДНК-наноструктур с помощью этой методики похожа на строительство моделей с помощью конструктора LEGO.
Нетривиальная идея о том, что из ДНК можно создавать сложные наноструктуры, была высказана еще в начале 1980‑х годов американским ученым Надрианом Симаном (Nadrian C. Seeman). Долгое время идея считалась совершенно утопической, пока в 2006 году молодой специалист по компьютерным наукам Пол Ротмунд (Paul W.K. Rothemund) не предложил оригинальную методику под названием «ДНК-оригами» (Paul W. K. Rothemund, 2006. Folding DNA to create nanoscale shapes and patterns), которая позволяла укладывать длинную одноцепочечную молекулу ДНК произвольным образом, соединяя ее в нужных участках «скрепками» — короткими ДНК-последовательностями, комплементарными этим нужным участкам (подробнее о методике рассказано в вышеупомянутой новости Елены Наймарк). С помощью ДНК-оригами было сложено множество разнообразных двухмерных и трехмерных фигур, в том числе смайлик размером 100 нм (рис.1).

- Рис. 1. Вот какие плоские структуры получил Пол Ротмунд с помощью методики ДНК-оригами. Изображение из статьи Paul W. K. Rothemund, 2006.
Однако, как ни была остроумна и перспективна эта методика, у нее был ряд серьезных недостатков. Во-первых, каждую новую структуру приходилось с нуля просчитывать заново: продумывать взаимное положение изгибов, подбирать нужные ДНК-последовательности, следить, чтобы скрепки были комплементарны только нужным участкам и не прилипали к чему-нибудь ненужному, и так далее. Еще одно затруднение возникало с основной нитью ДНК: если коротенькие скрепки можно было без проблем синтезировать искусственно, то синтез с нуля мало-мальски длинной нуклеотидной последовательности — дело трудное, долгое и неблагодарное: чем длинней нить, тем больше вероятность, что она будет синтезирована с ошибкой. Поэтому обычно в качестве основной нити в ДНК-оригами используется уже готовая ДНК — например, принадлежащая какому-нибудь вирусу; а это, опять же, накладывает на методику определенные ограничения. К тому же, оригами-структуры долго собираются (видимо, из-за того, что нужно укладывать длинную — а значит, легко запутывающуюся — нить ДНК), и выход их невелик.
Поэтому необходимо было развивать и другие пути получения сложных конструкций из ДНК. Например, чрезвычайно соблазнительно выглядела идея создания модульных ДНК-структур, то есть таких, которые состоят из маленьких «кирпичиков» — олигонуклеотидных кусочков, налипающих друг на друга благодаря своей комплементарности и таким образом собирающихся в сложные структуры. Такие маленькие кусочки легко синтезировать, их сборка должна отнимать меньше времени, чем оригами, а складывать из них можно самые разные фигуры.
На первый взгляд идея кажется очень простой, однако в ней есть гигантский подводный камень: ДНК — это, как-никак, двойная спираль, и если модульный «кирпичик» будет недостаточно продуман, то спирали, образованные разными модулями, станут налезать друг на друга, не позволяя итоговой структуре правильно собраться.
Первая, еще несовершенная, работа, показывающая потенциал модульных ДНК-структур, была опубликована в 2003 году (Hao Yan et al, 2003. DNA-Templated Self-Assembly of Protein Arrays and Highly Conductive Nanowires). Модули представляли собой что-то вроде крестообразных черепиц, которые можно было укладывать в плоские решетки или ленты (рис. 2). Авторы постарались придать своим черепичкам как можно более стабильную пространственную конфигурацию, что, по нынешним временам, оказалось стратегической ошибкой: во-первых, черепички надо было долго и кропотливо синтезировать, а во-вторых, они из-за своей «неповоротливости» могли образовывать только плоские и однообразные структуры.
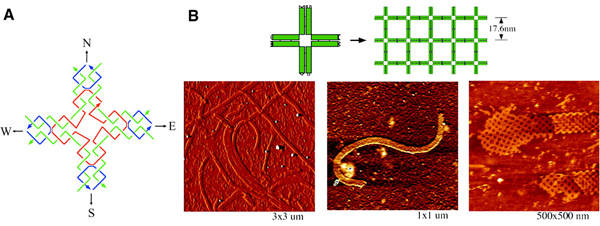
- Рис. 2. Крестообразная ДНК-черепица состоит из нескольких сложно переплетенных нитей ДНК и может дать начало плоским структурам. Фотографии этих структур справа внизу получены с помощью сканирующего атомного силового микроскопа на разных разрешениях и в разных режимах. Изображение из статьи Hao Yan et al, 2003.
Пять лет спустя, в 2008 году, был предложен еще один вариант ДНК‑модуля (Peng Yin et al, 2008. Programming DNA Tube Circumferences). Это тоже было что-то вроде черепицы, но гораздо более интересной. Она была проще по строению и представляла собой, по сути, просто короткую (всего лишь 42‑нуклеотидную) и потому легкосинтезируемую нить ДНК. Эта нить подразделялась на четыре домена, на каждый из которых мог налипнуть домен соседней нити. В результате черепица особым образом изгибалась и самособиралась в плоские структуры, которые могли замыкаться в трубки (рис. 3).
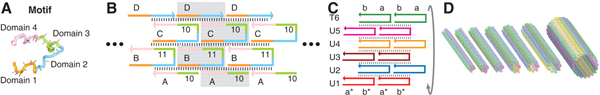
- Рис. 3. Изогнутая ДНК-черепица (A) и получаемые из нее плоские структуры (B), которые при наличии «липких» комплементарных концов (a и a*, b и b*) сворачиваются (C) и образуют трубки (D). Изображение из статьи Peng Yin et al, 2008
Но и трехмерные трубки — это тоже было слишком мало: ученых снедала мечта о полноценных, произвольных трехмерных структурах, которые можно было бы легко собрать из простых модулей.
И вот Пэн Инь, первый автор из предыдущей работы, став руководителем собственной группы, выпустил новую статью, в которой предложил еще один вид ДНК-модуля, напоминающий кирпичик из детской игры LEGO (рис. 4). Этот «кирпичик» очень похож на «черепичку» из предыдущей работы: это тоже нить, имеющая четыре «липких» домена для присоединения комплементарных участков других нитей, но еще более короткая (всего 32 нуклеотида) и немного иначе изогнутая. В отличие от черепиц, кирпичи при склеивании поворачиваются друг относительно друга на 90°. Этот поворот дает целых две больших выгоды: во-первых, благодаря нему структура становится объемной, а во-вторых, поскольку 90° — это четверть полного, 360-градусного поворота, то каждый пятый кирпич в ряду будет принимать то же положение, что и первый, и двойные спирали, образуемые разными модулями, не будут друг на друга наезжать, а четко и аккуратно выстроятся в пространстве.
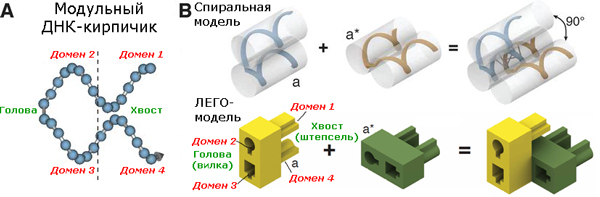
- ДНК-кирпичик и получаемые из него объемные структуры. Каждый 32‑нуклеотидный кирпичик имеет четыре «липких домена», которые образуют участки двойной спирали с комплементарными доменами других кирпичиков. Кирпичик состоит из двух 16‑нуклеотидных антипараллельных спиралей, соединенных между собой одиночной фосфатной связью, в результате чего приобретает форму, отдаленно напоминающую форму двойного кирпичика лего, где два «хвостовых» домена аналогичны лего-штепселю, а два «головных» — лего-розетке. Соединяясь между собой, кирпичики поворачиваются друг относительно друга на 90°, что позволяет им образовывать трехмерные структуры. При этом, каждый домен каждого кирпичика может соединиться только с одним, комплементарным себе, доменом другого кирпичика. Изображение из обсуждаемой статьи в Science, с изменениями
В результате если мы один раз просчитаем набор кирпичиков для параллелепипеда достаточно большого объема, то руки у нас развязаны: мы сможем собрать из этого набора почти любую объемную структуру, которая помещается внутри параллелепипеда. Однако надо помнить, что минимальная объемная точка (воксел) в этой структуре не может быть меньше, чем объем витка двойной спирали, образованной комплементарными доменами двух кирпичиков, который составляет 2,5 × 2,5 × 2,7 нм. Это накладывает определенные ограничения на построение объемных моделей, но пока, на данном этапе развития нанотехнологий, эти ограничения не кажутся особенно вопиющими.
Исследователи наигрались вволю, построив из своих кубиков более ста различных объемных структур, в том числе модель космического шаттла и пронзенное сердце. Почти все модели сложились правильно, показав не худший (а то и лучший) выход, чем при методике ДНК-оригами, однако у семи моделей возникли различные проблемы со сборкой, уровнем выхода или стабильностью конструкции. Впрочем, тот, кто играл в детстве в «Лего», наверное, помнит, что очень сложные формы с далеко выступающими частями из этого конструктора получаются плохо.
Итак, подытожим. Перед нами остроумная, простая, гибкая и неприхотливая методика, которая не требует ни экзотических условий, ни дорогих реактивов и оборудования. По сути дела, всё, что нам нужно сделать, — это просто добавить нужные кирпичики в специально подготовленный раствор и отжечь их (см. отжиг), чтобы комплементарные домены соединились между собой. Хотя некоторые пространственные ограничения накладывает строгий размер воксела, но и эту проблему можно обойти, если использовать кирпичики с измененными свойствами (исследователи немного продвинулись в этом направлении, создав, например, трехмерную модель, напоминающую медовые соты, из кирпичиков другой конфигурации, которые соединялись с поворотом в 120°). Еще одно ограничение — нестабильность особенно сложных структур — можно обойти, скомпилировав лего-методику с методикой ДНК-оригами: в тех участках, где кирпичи разваливаются, не в силах поддержать сложную форму, можно использовать длинную оригамную нить, а прочие части структуры оставить «кирпичными».
Впечатляет и возможный размер лего-моделей. В идеале (если использовать все возможные кирпичи) он может составить до 524 288 нуклеотидов, и даже этот размер можно превысить, если собирать структуру в несколько этапов. Хотя пока что особенно крупные лего-структуры получаются нестабильными, авторы предложили несколько методов решения этой проблемы, которые, возможно, позволят довести возможный размер моделей до астрономических (в наномасштабе, конечно) величин.
Возможности применения для таких ДНК-структур огромны, начиная от использования в наноэлектрических приборах и заканчивая доставкой лекарств. Одним словом, совершенно ясно, что лего-ДНК-модели вот-вот получат практическое применение и принесут человечеству немалую пользу.
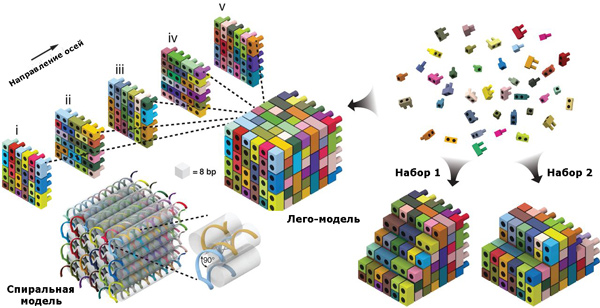
- Рис. 5. Если у нас есть набор кирпичиков, из которого можно собрать параллелепипед, то потом из этого набора можно получить и другие фигуры, выбирая только нужные кирпичики. Изображение из обсуждаемой статьи в Science
Текст: Вера Башмакова
Источник: Элементы
Комментарии
Комментарии
(от лат. armilla - кольцо) Настольное приспособление, состоящее из подвижных обручей и дисков. Оно предназначено для наглядного представления небесной сферы, а также точек, линий и плоскостей, которые... [далее]
Сайт разработан и поддерживается лабораторией 801 Института космических исследований Российской академии наук.
Подбор материалов - Н.Санько
Полное или частичное использование размещённых на сайте материалов
возможно только с обязательной ссылкой на сайт Секция Солнечная система Совета РАН по космосу.